近日,中南大学湘雅药学院曹东升副教授团队在国际生物信息学权威期刊《Briefings in Bioinformatics》(IF=9.101, JCR1区Top期刊)以论著形式在线发表了题为“Improving Structure-Based Virtual Screening Performance via Learning From Scoring Function Components”(DOI:10.1093/bib/bbaa094)的最新研究成果。该研究提出了一种借助机器学习算法改善对接软件筛选能力的方法。
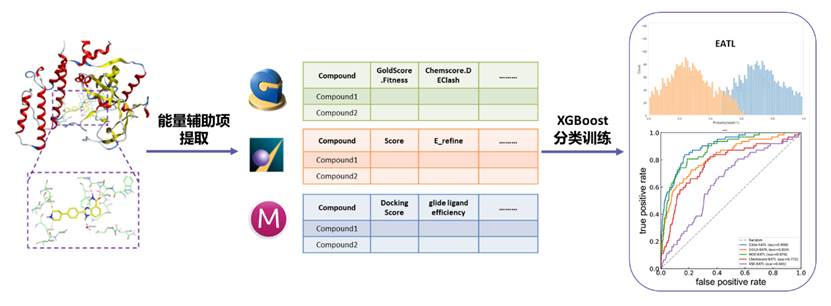
分子对接模拟是基于结构药物设计的重要手段。蛋白配体对接的一般过程是把配体小分子放在受体的活性口袋中,通过不断优化小分子的位置以及分子内部柔性键的二面角,寻找小分子化合物和靶标大分子相互作用的最佳构象,然后使用打分函数计算其相互作用和结合能。传统的打分函数通常选取专家挑选的少量特征与实验测定亲和力值进行线性回归,但是这种线性关系并不总是存在的,因而无法满足实际使用需求。提升对接打分函数的虚拟筛选能力,能够极大地节约时间和资源成本,加速先导化合物的发现过程。
本研究提取了MOE, GOLD,Schrödinger三个对接软件共计61种能量辅助项作为蛋白-配体相互作用的表征,使用XGBoost算法进行分类模型训练,预测小分子化合物与目标靶点结合的可能性,从而提升对接软件的虚拟筛选性能。该方法在8个多样性靶点数据集上进行验证,使用多种虚拟筛选指标(ROC, BEDROC, logAUC, EF)进行评估,证实其能够显著提升基于对接的虚拟筛选的效率并且优于其他同类型的筛选辅助模型。该方法操作简便,将经验知识与机器学习相结合,可以推广到其他能够提供能量辅助项的对接软件中。
湘雅药学院2019级硕士研究生熊国丽为本文第一作者,曹东升副教授与浙江大学侯廷军教授为通讯作者。另外,系列分子对接算法研究近期发表于《Briefings in Bioinformatics》(IF = 9.101,2篇),《Journal of Chemical Information and Modeling》(IF = 3.966,1篇),《Journal of Chemical Theory and Computation》(IF = 5.313,1篇),《Physical Chemistry Chemical Physics》(IF = 3.520,1篇),通讯作者为曹东升副教授和浙江大学侯廷军教授。上述工作得到了国家重点基础研究计划青年科学家项目,国家自然科学基金的支持。
